Veel imkers die zich bezighouden met het telen van de zwarte bij, maken daarbij gebruik van uiterlijke kenmerken van de werksters om te oordelen over de zuiverheid van hun bijenvolken. In dat kader is het onderzoek van de vleugelmorfometrie het meest populair. Doorheen de afgelopen decennia zijn daaromtrent dan ook verschillende programma’s verschenen: CBeeWing, Flügel-Index, IdentiFly, DeepWings, … Echter, men gaat vaak volledig voorbij aan de vraag of vleugelmorfometrie in hun geval wel zinvol is. Om die vraag te kunnen beantwoorden, moet gekeken worden naar wat vleugelmorfometrie ons wel en niet kan vertellen.
De studie van uiterlijke kenmerken van honingbijen gaat terug naar de 18de en 19de eeuw, toen men honingbijen van over heel Europa uitgebreid begon te bestuderen. Men stelde daarbij vast dat honingbijen in Europa er niet overal hetzelfde uitzagen, noch hetzelfde gedroegen. Dat leidde tot het besluit dat er in Europa verschillende ondersoorten van de honingbij leven. Makkelijk observeerbare kenmerken, zoals vleugelmorfometrie, werden zo handige instrumenten om zuivere ondersoorten van elkaar te onderscheiden en een willekeurige honingbij, behorend tot een van de gekende zuivere ondersoorten, op naam te brengen (identificeren).

Vele decennia later zijn imkers die identificatietechnieken beginnen gebruiken om de zuiverheid van hun bijenvolken te kunnen bepalen. Maar … Zowel zwak als sterk gehybridiseerde bijenvolken hebben voor die kenmerken vaak zeer uiteenlopende waarden waar overigens helemaal geen referentie voor bestaat. Een willekeurige gehybridiseerde honingbij kan op die manier plots voldoen aan de referentiewaarden voor eender welke zuivere ondersoort of zelfs aan geen van alle referentiewaarden. Dit heeft als gevolg dat uiterlijke kenmerken, zoals vleugelmorfometrie, compleet betekenisloos worden zodra ook maar een weinig hybridisatie in de populatie is opgetreden. Anders gezegd: imkers zijn de identificatietechnieken verkeerdelijk gaan gebruiken voor een toepassing waar die technieken nooit voor bedoeld waren en ook gewoon niet voor gebruikt kunnen worden.
Vleugelmorfometrie kort samengevat:
- Voldoet de vleugelmorfometrie van een honingbij NIET aan de referentiewaarden van de zwarte bij, dan is deze honingbij gehybridiseerd, MAAR is de mate van hybridisatie nog steeds ongekend: zowel zwakke als sterke hybridisatie is mogelijk.
- Voldoet de vleugelmorfometrie van een honingbij WEL aan de referentiewaarden van de zwarte bij, dan is deze honingbij een zuivere zwarte bij ENKEL OP VOORWAARDE DAT de honingbij afkomstig is van een genetisch zuivere populatie zwarte bijen.
Helaas zijn de meeste resterende populaties zwarte bijen lichtjes genetisch vervuild (zo ook Chimay, Texel en Terschelling) waardoor goede waarden voor vleugelmorfometrie geen garantie meer zijn voor zuivere bijenvolken. Met andere woorden, betrekt men teeltmateriaal van een populatie waar ooit enige hybridisatie (hoe miniem ook) opgetreden heeft, dan kan men niet op vleugelmorfometrie vertrouwen om de zuiverheid te monitoren. Gelukkig is er een andere methodiek waarmee de zuiverheid wél op betrouwbare wijze kan worden vastgesteld: analyse van het nucleair DNA1 met behulp van SNPs (Single Nucleotide Polymorphism), zogenaamde puntmutaties in het DNA.
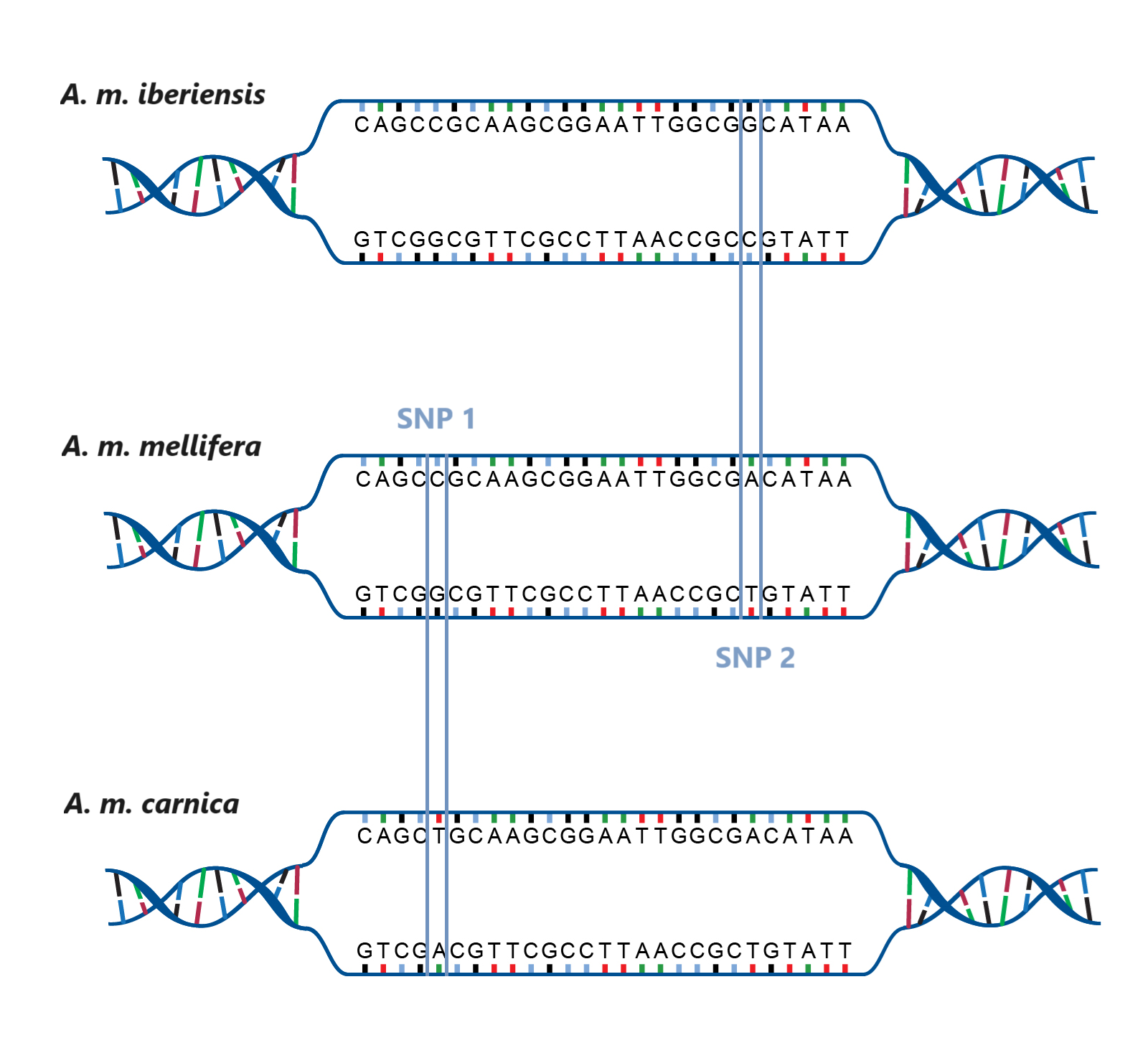
Los van het gegeven dat via DNA de genetische zuiverheid rechtstreeks wordt onderzocht (uiterlijke kenmerken worden immers indirect door het DNA bepaald), heeft deze methodiek nog 2 zeer krachtige voordelen in vergelijking met voornoemde vleugelmorfometrie:
- Uitgebreide set aan meetpunten: waar bij vleugelmorfometrie de hele analyse gevoerd wordt op basis van maximaal 19 meetpunten (zijnde alle knooppunten van vleugeladers), is de hoeveelheid meetpunten bij DNA-analyse (SNPs) vele malen groter. Zo hanteren we bij Imkerij Merodegoud de Illumina HONEYBEE2021 chip die een analyse met maar liefst 70 814 SNPs toelaat.
- Uitgebreid referentiemateriaal: waar de referentiewaarden voor vleugelmorfometrie gebaseerd zijn op slechts zeer weinig bijenvolken (voor bv. IdentiFly slechts 14 bijenvolken van de zwarte bij!), werden de SNPs geselecteerd door analyse van zwarte bijen van honderden bijenvolken afkomstig uit heel West- en Noord-Europa.
DNA-analyse met SNPs stelt ons zo in staat om een zeer precieze en nauwkeurige inschatting te maken van de genetische zuiverheid van honingbijen. Let wel, het gaat over een inschatting: de DNA-analyse geeft als resultaat de waarschijnlijkheid (de kans) dat de geteste honingbij tot de ene of de andere ondersoort behoort. Op internationaal niveau hebben wetenschappers de consensus opgemaakt dat de ondergrens op 90% ligt: van een honingbij die op basis van DNA-analyse minstens 90% kans heeft om een zwarte bij te zijn, wordt aangenomen dat het effectief een zwarte bij is.
De reden waarom het over een inschatting (waarschijnlijkheid) gaat, is omdat het nooit uitgesloten is dat een SNP, die initieel als ondersoort-specifiek geïdentificeerd werd, uiteindelijk toch geen ondersoort-specifieke SNP is. Dit heeft te maken met de beperking op referentiemateriaal. Het is immers onmogelijk om van elk bijenvolk in Europa een staal te nemen om ondersoort-specifieke SNPs te zoeken. De 90%-ondergrens houdt daarom preventief rekening met dat risico.
Een lang verhaal in het kort: wil men op beide oren kunnen slapen betreft de zuiverheid van zijn/haar bijenvolken, dan kiest men anno 2024 vanzelfsprekend voor de analyse van nucleair DNA met SNPs.

- Het nucleair DNA (ncDNA) is dat DNA welk in de celkern zit en waarvan de ene helft geërfd wordt van de moeder (koningin) en de andere helft van de vader (dar). Dit ncDNA beïnvloedt de haarkleur, de vleugelmorfometrie, de neiging tot propoliseren, de zachtaardigheid, … Naast het ncDNA bezitten dieren ook mitochondriaal DNA (mtDNA) dat in de mitochondriën zit en uitsluitend langs moeders kant wordt overgedragen. mtDNA is zeer beperkt in hoeveelheid en heeft enkel betrekking op de stofwisseling van het organisme, maar kan omwille van de erfbaarheid langs moeders zijde wel gebruikt worden om de evolutionaire linie (M, C, O, A, Y) te achterhalen. In het kader van conservatie is ncDNA dus veel belangrijker dan mtDNA. ↩︎
- Henriques, D., Browne, K.A., Barnett, M.W. et al. High sample throughput genotyping for estimating C-lineage introgression in the dark honeybee: an accurate and cost-effective SNP-based tool. Sci Rep 8, 8552 (2018). https://doi.org/10.1038/s41598-018-26932-1 ↩︎

