Viele Imker, die sich mit der Zucht der Dunklen Biene beschäftigen, nutzen äußere Merkmale der Arbeiterinnen, um die Reinheit ihrer Bienenvölker zu beurteilen. In diesem Zusammenhang ist die Untersuchung der Flügelmorphometrie am beliebtesten. In den vergangenen Jahrzehnten sind daher verschiedene Programme erschienen: CBeeWing, Flügel-Index, IdentiFly, DeepWings usw. Allerdings wird oft die Frage übersehen, ob die Flügelmorphometrie in ihrem Fall sinnvoll ist. Um diese Frage beantworten zu können, muss man betrachten, was die Flügelmorphometrie uns sagen kann und was nicht.
Die Erforschung äußerer Merkmale von Honigbienen reicht zurück ins 18. und 19. Jahrhundert, als man begann, Honigbienen aus ganz Europa ausführlich zu studieren. Dabei stellte man fest, dass Honigbienen in Europa nicht überall gleich aussahen oder sich gleich verhielten. Dies führte zur Erkenntnis, dass es in Europa verschiedene Unterarten der Honigbiene gibt. Leicht beobachtbare Merkmale wie die Flügelmorphometrie wurden so zu nützlichen Werkzeugen, um reine Unterarten voneinander zu unterscheiden und eine zufällige Honigbiene, die zu einer der bekannten reinen Unterarten gehört, zu identifizieren.

Die rechte Vorderflügel einer Honigbiene, die Knoten der Flügeladern bilden die Grundlage für die Flügelmorphometrie
Einige Jahrzehnte später begannen Imker, Identifikationstechniken zu verwenden, um die Reinheit ihrer Bienenvölker zu bestimmen. Aber… Sowohl schwach als auch stark hybridisierte Bienenvölker zeigen oft sehr unterschiedliche Werte für diese Merkmale, für die es außerdem überhaupt keine Referenz gibt. Eine zufällig hybridisierte Honigbiene kann auf diese Weise plötzlich den Referenzwerten für jede reine Unterart entsprechen oder sogar überhaupt keinen der Referenzwerte erfüllen. Dies hat zur Folge, dass äußere Merkmale wie die Flügelmorphometrie vollkommen bedeutungslos werden, sobald auch nur eine geringe Hybridisierung in der Population auftritt. Anders ausgedrückt: Imker haben die Identifikationstechniken falsch für eine Anwendung eingesetzt, für die diese Techniken nie gedacht waren und auch einfach nicht verwendet werden können.
Flügelmorphometrie kurz zusammengefasst:
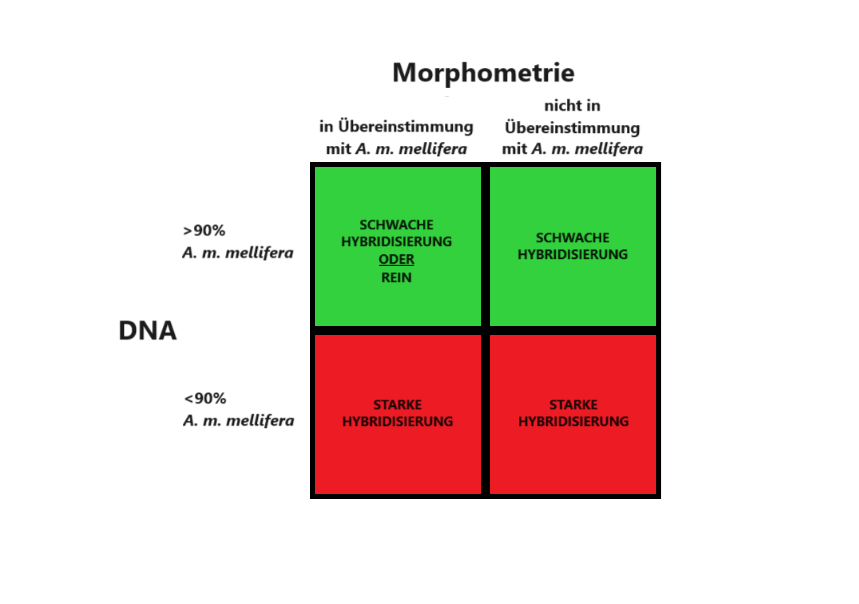
- Erfüllt die Flügelmorphometrie einer Honigbiene NICHT die Referenzwerte der Dunklen Biene, dann ist diese Honigbiene hybridisiert, ABER der Grad der Hybridisierung ist immer noch unbekannt: Sowohl schwache als auch starke Hybridisierung sind möglich.
- Erfüllt die Flügelmorphometrie einer Honigbiene WOHL die Referenzwerte der Dunklen Biene, dann handelt es sich bei dieser Honigbiene um eine reine Dunkle Biene, NUR UNTER DER BEDINGUNG, dass die Honigbiene aus einer genetisch reinen Population von Dunklen Bienen stammt.
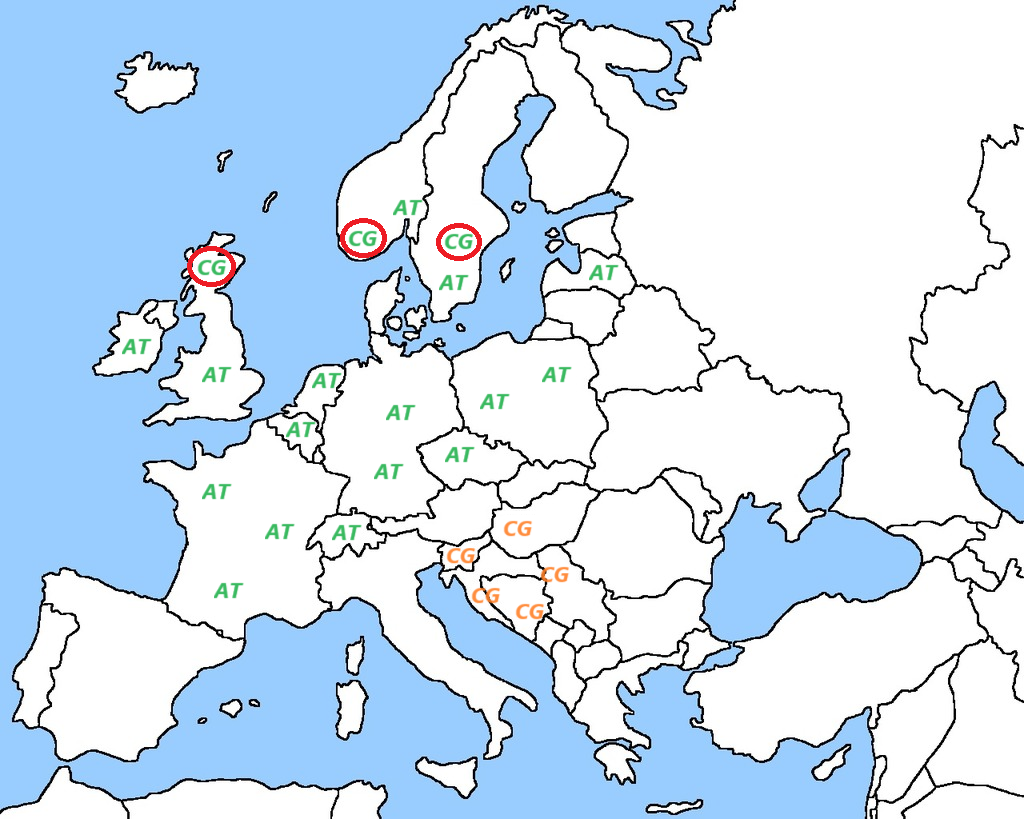
Leider sind die meisten verbleibenden Populationen von Dunklen Bienen leicht genetisch verunreinigt (wie auch in Belgien, Niederlande, Norwegen, Schweden, Schweiz usw.), wodurch gute Werte für die Flügelmorphometrie keine Garantie mehr sind für reine Bienenvölker. Anders ausgedrückt, wenn man Zuchtstoff aus einer Population bezieht, in der jemals eine Hybridisierung (wie gering auch immer) stattgefunden hat, kann man sich nicht auf die Flügelmorphometrie verlassen, um die Reinheit zu überwachen. Glücklicherweise gibt es eine andere Methodik, mit der die Reinheit auf zuverlässige Weise festgestellt werden kann: die Analyse der Kern-DNA1 unter Verwendung von SNPs (Single Nucleotide Polymorphism), sogenannten Punktmutationen in der DNA.
Abgesehen davon, dass durch DNA die genetische Reinheit direkt untersucht wird (da äußere Merkmale indirekt durch einige Gene bestimmt werden), hat diese Methodik noch zwei sehr starke Vorteile im Vergleich zur den Flügelmorphometrie:
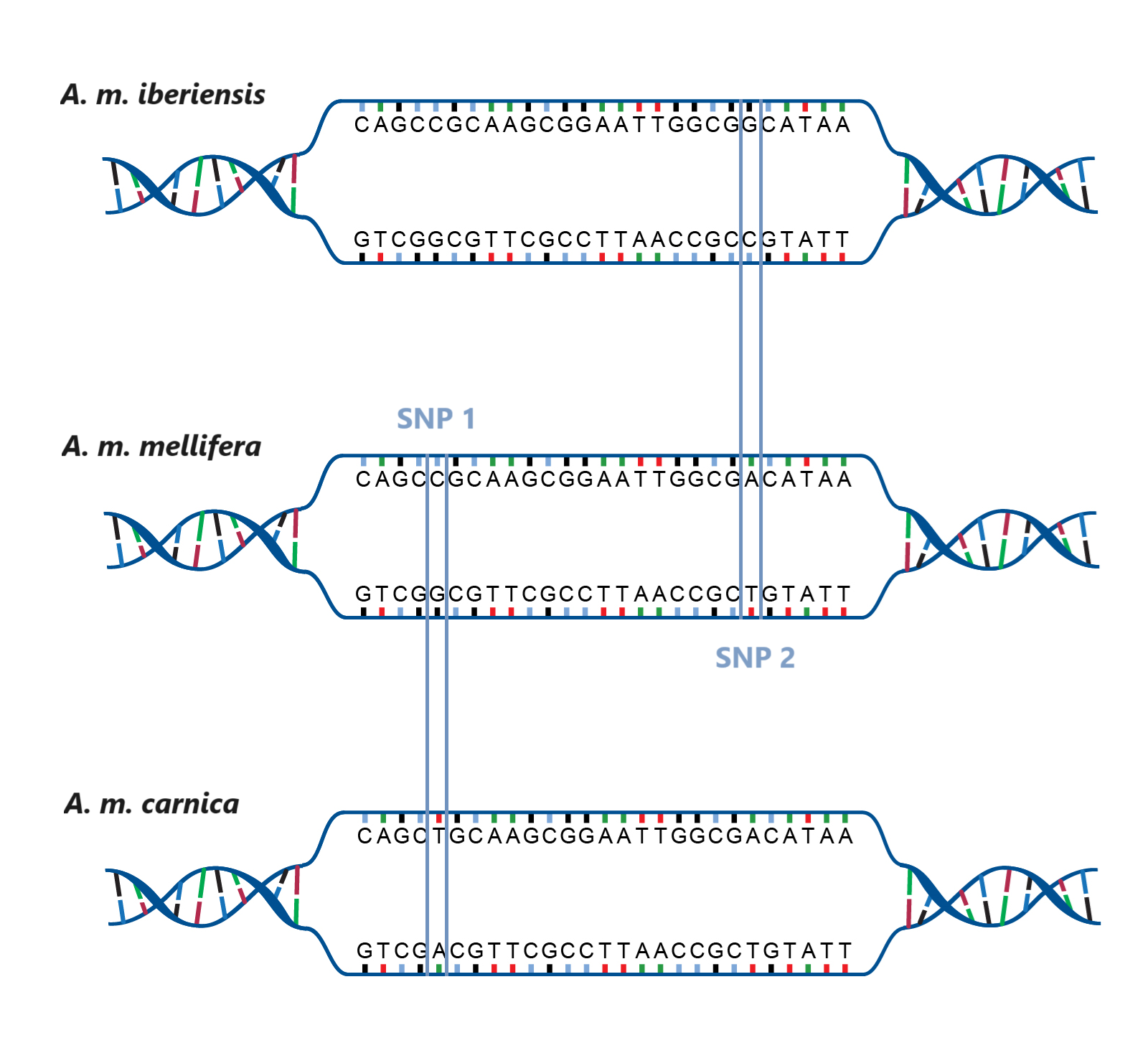
- Umfangreicher Satz an Messpunkten: Während bei der Flügelmorphometrie die gesamte Analyse auf maximal 19 Messpunkten basiert (alle Knoten der Flügeladern), ist die Anzahl der Messpunkte bei der DNA-Analyse (SNPs) um ein Vielfaches größer. Bei Imkerei Merodegoud verwenden wir den Illumina HONEYBEE2021 Chip, der eine Analyse mit 70.814 SNPs ermöglicht.
- Umfangreiches Referenzmaterial: Während die Referenzwerte für die Flügelmorphometrie auf nur sehr wenigen Bienenvölkern basieren (zum Beispiel verwendet IdentiFly nur 14 Bienenvölker der Dunklen Biene!), wurden die SNPs durch die Analyse von Dunklen Bienen aus Hunderten von Bienenvölkern aus ganz West- und Nordeuropa ausgewählt.
Die DNA-Analyse mit SNPs ermöglicht es uns, eine sehr präzise und genaue Einschätzung der genetischen Reinheit von Honigbienen vorzunehmen. Beachten Sie jedoch, dass es sich um eine Einschätzung handelt: Die DNA-Analyse liefert das Ergebnis der Wahrscheinlichkeit (der Chance), dass die getestete Honigbiene zu einer bestimmten Unterart gehört oder nicht. Auf internationaler Ebene haben Wissenschaftler den Konsens gefunden, dass die Untergrenze bei 90% liegt: Eine Honigbiene, die aufgrund der DNA-Analyse mindestens 90% Wahrscheinlichkeit hat, eine Dunkle Biene zu sein, wird als tatsächlich eine Dunkle Biene betrachtet.
Der Grund, warum es sich um eine Einschätzung (Wahrscheinlichkeit) handelt, liegt darin, dass nie ausgeschlossen werden kann, dass ein SNP, der ursprünglich als unterartspezifisch identifiziert wurde, letztendlich doch kein unterartspezifischer SNP ist. Dies hängt mit den Einschränkungen des Referenzmaterials zusammen. Es ist nämlich unmöglich, von jeder Bienenvolk und Museumssammlung in Europa eine Probe zu entnehmen, um unterartspezifische SNPs zu suchen. Die 90%-Untergrenze berücksichtigt daher vorsorglich dieses Risiko.
Kurz gesagt: Um beruhigt über die Reinheit seiner Bienenvölker zu sein, wählt man im Jahr 2024 natürlich die Analyse von Kern-DNA mit SNPs.
- Kern-DNA (ncDNA) ist das DNA, das sich im Zellkern befindet und von dem eine Hälfte von der Mutter (Königin) und die andere Hälfte vom Vater (Drohn) geerbt wird. Dieses ncDNA beeinflusst die Haarfarbe, die Flügelmorphometrie, die Tendenz zur Propolisierung, die Sanftmut, usw. Neben dem ncDNA besitzen Tiere auch mitochondriales DNA (mtDNA), das sich in den Mitochondrien befindet und ausschließlich über die mütterliche Linie übertragen wird. mtDNA ist in seiner Menge sehr begrenzt und betrifft nur den Stoffwechsel des Organismus, kann jedoch aufgrund der Vererbbarkeit über die mütterliche Seite verwendet werden, um die evolutionäre Linie (M, C, O, A, Y) zu verfolgen. Im Rahmen der Erhalt der Unterarten ist ncDNA also viel wichtiger als mtDNA. ↩︎
- Henriques, D., Browne, K.A., Barnett, M.W. et al. High sample throughput genotyping for estimating C-lineage introgression in the dark honeybee: an accurate and cost-effective SNP-based tool. Sci Rep 8, 8552 (2018). https://doi.org/10.1038/s41598-018-26932-1 ↩︎

